<はじパタ> 10.4 確率モデルによるクラスタリング
はい、ここまで10章の教師なし学習のクラスタリングをやってまいりましたが、10章も最後となりました。とりあえず一章をきちんと走り抜けられてよかったです。 で、ここは前回までのK-平均法や融合法と違い、各要素がそれぞれのクラスに入る確率を求める確率モデルによるクラスタリングをおこないます。キーワードとしては、混合正規分布モデルとEMアルゴリズムとなるようです。わかりやすい説明はこちらのリンクをご参照、とりあえず自分なりにまとめてみます。
混合正規分布モデル
各クラスタの分布がそれぞれ正規分布にしたがうと仮定をしてその平均と分散が一番Fitする最尤の値を求めていくものになります。また、ある要素に関してそれぞれクラスタの割合を表現する混合比なる重みづけのためのパラメータもでてきてこの値もいじる必要があります。
EMアルゴリズム
所属しているクラスタを示す隠れ変数があるものとして、その変数の確率モデルの最尤推定値を求めるのに優れているのが、EMアルゴリズムとなるようです。E(Expectation)ステップで隠れ変数に対しての期待値を計算、M(Maximization)ステップでその隠れ変数の最大値を計算するようにします。
はじパタに限らず、いくつかの本にこのEMのステップで何が行われているかの数式や説明があるのですが、いかに示すサイトの説明が図として直感的に何をしているのかわかりやすく書かれていると思いました。こういうの貴重。
www.slideshare.net
ピーナッツを縦に割っちゃう問題
それで、コーディングを走らせてみたのですが、どうしても正解率が上がらないんです。実際にプロットをしてみると以下のように右上のピーナッツを縦にわるようなクラスタリングをしてしまうのです。

これは初期値にだいぶよるようですね。平均値のmuの値を意図的に変えてみたところ以下のようにピーナッツを横にわるような「理想的な」クラスタに収束することもあるようです。
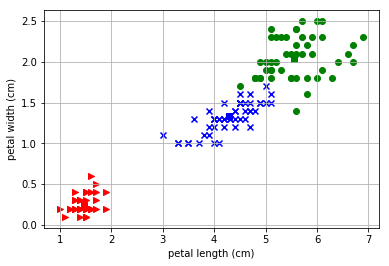
初期値をランダムに置くようにして確認してみましたが、この理想的な形状に収まるのは10回に1~2回程度のようです。
合わないモデル?
初期値問題に頭を抱えて機械学習のお師匠様のところに相談したところ、「そもそも合わないモデルだったのでは」とのことです。考察としてはアヤメデータ以外でもこのEMアルゴリズムを試してみてうまく当てはまるかを確認してみたほうが良いかもしれないです。やるかはわからないですが。
ソースコード
いつも以上にとっちらかったソースになってしまっていると思います。まあ備忘録なので。
import numpy as np from sklearn import datasets import itertools import matplotlib.pyplot as plt #inline pyplot iris = datasets.load_iris() features = iris.data feature_names = iris.feature_names targets = iris.target from collections import Counter class Iris_PModel: def __init__(self): self.p = [] self.x = [] self.y = [] self.t = [] self.pt = [] self.num = [50, 50, 50] iris = datasets.load_iris() features = iris.data targets = iris.target def install(self, a=2, b=3): self.x = features.T[a] self.y = features.T[b] self.t = targets self.pi = [1/3, 1/3, 1/3] self.means_dists() def plot_map(self, df=False): p = self.pt if df: p = self.t for t, marker, c in zip(range(3), '>ox', 'rgb'): plt.scatter( features[p == t, 2], features[p == t, 3], marker=marker, c=c, ) plt.scatter( self.mu[t][0], self.mu[t][1], marker='s', c=c ) plt.xlim(1.9, 4.5) plt.ylim(0.9, 7.1) plt.xlabel(feature_names[2]) plt.ylabel(feature_names[3]) plt.autoscale() plt.grid() plt.show() def randomize(self): self.pt = np.array([np.random.randint(3) for i in range(sum(self.num))]) def norm(self, mu, sigma): n_mu = np.array(mu) n_sig = np.array([[sigma[0], sigma[2]],[sigma[2], sigma[1]]]) n_det = np.linalg.det(n_sig) n_inv = np.linalg.inv(n_sig) nv = [] for i in range(150): n_c = np.array([self.x[i], self.y[i]]) - n_mu nv.append((1/(2*np.pi*np.sqrt(n_det))) * np.exp(- n_c.dot(n_inv).dot(n_c).T/2)) return(nv) def means_dists(self, init=True): if init: self.randomize() x_m = sum(self.x)/150 y_m = sum(self.y)/150 self.nu = [[np.random.normal(x_m, 1), np.random.normal(y_m, 1)], [np.random.normal(x_m, 1), np.random.normal(y_m, 1)], [np.random.normal(x_m, 1), np.random.normal(y_m, 1)]] print("mu", self.mu) self.v = [[1.0, 1.0, 0.0], [1.0, 1.0, 0.0], [1.0, 1.0, 0.0]] def m_step(self, gamma): n_k = [sum([gamma[i][j] for i in range(150)]) for j in range(3)] self.mu = [[sum([self.x[i]*gamma[i][j] for i in range(150)])/n_k[j], sum([self.y[i]*gamma[i][j] for i in range(150)])/n_k[j]] for j in range(3)] self.v = [[sum([(self.x[i]-self.mu[j][0])**2*gamma[i][j] for i in range(150)])/n_k[j], sum([(self.y[i]-self.mu[j][1])**2*gamma[i][j] for i in range(150)])/n_k[j], sum([(self.x[i]-self.mu[j][0])*(self.y[i]-self.mu[j][1])*gamma[i][j] for i in range(150)])/n_k[j]] for j in range(3)] self.pi = [n_k[i]/150 for i in range(3)] def e_step(self, init=False): if init: self.install(2, 3) nv = [self.norm( self.mu[i], self.v[i]) for i in range(3)] snv = [nv[0][i]*self.pi[0] + nv[1][i]*self.pi[1] + nv[2][i]*self.pi[2] for i in range(150)] prop = [[nv[0][i]*self.pi[0]/snv[i], nv[1][i]*self.pi[1]/snv[i], nv[2][i]*self.pi[2]/snv[i]] for i in range(150)] return prop def process(self): gamma = self.e_step(True) self.m_step(gamma) pre_result = [] for i in range(31): result = [] for g in gamma: result.append(g.index(max(g))) self.pt = np.array(result) max_int = [0, 0, 0] if pre_result == result: print(self.pi) self.plot_map() break pre_result = list(result) gamma = self.e_step(False) self.m_step(gamma) krs = Iris_PModel() krs.process()